Simplificando análises de dados manuais do Excel usando o Python

Tratar e analisar dados no Excel pode ser um verdadeiro caos, mesmo que você precise fazer coisas simples como cruzar duas tabelas de dados. Uma solução melhor é o uso de scripts em Python, que possibilitam a automação de tarefas repetitivas e manuais. Neste artigo mostramos um exemplo simples, comparando o Excel versus Python.
Como automatizar o tratamento de dados feito no Excel usando o Python?
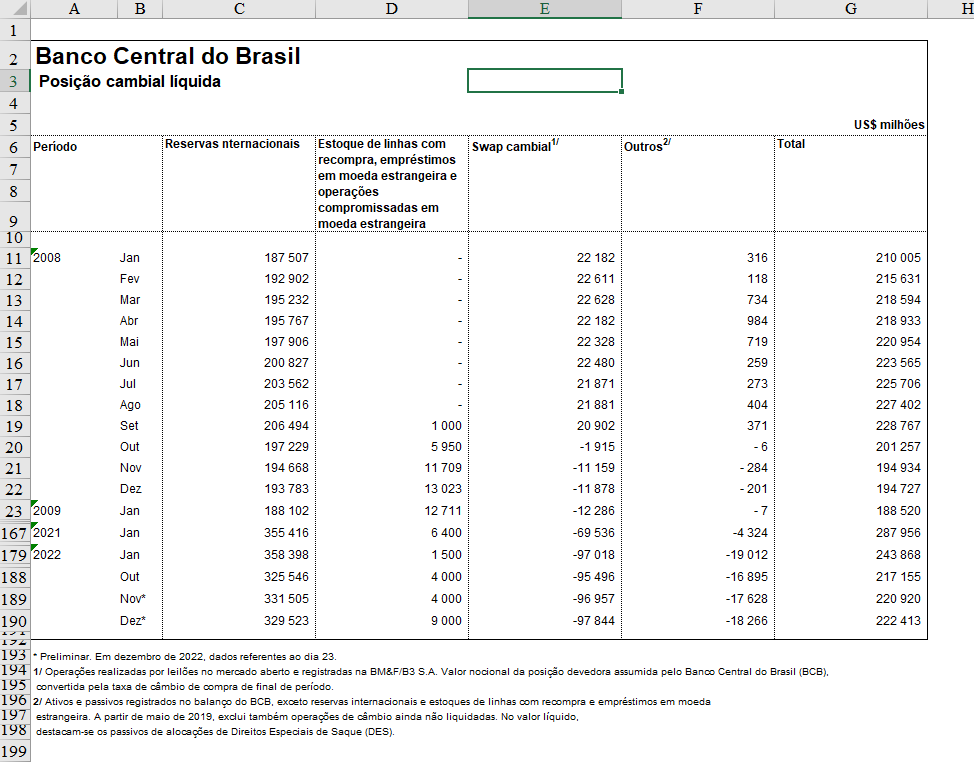
Segundo a pesquisa “State of Data Science”, profissionais de dados gastam 3 horas/dia (38% do tempo) apenas preparando os dados, antes mesmo de analisá-los. Neste artigo advogamos que este gasto de tempo pode ser drasticamente reduzido ao utilizar ferramentas open source, como Pandas e Python, para automatizar tarefas repetitivas que costumam ser feitas em Excel.
O que são APIs e como utilizá-las no Python?
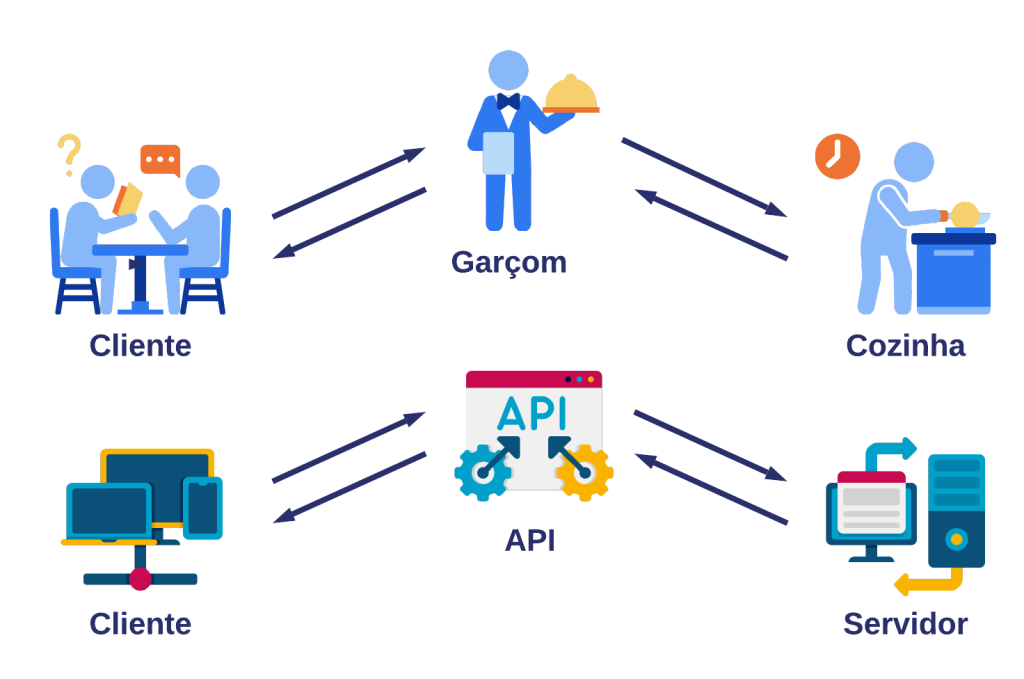
Utilizar APIs para coletar dados online pode acelerar e automatizar significativamente o processo de análise de dados. Diversas bases de dados hoje em dia possuem APIs para livre utilização, como Banco Central, Portal de Dados Abertos, IBGE e outras. Neste artigo, explicamos intuitivamente o que são APIs, como funcionam e mostramos um exemplo em Python de utilização.
Como fazer web scraping no Python? Um tutorial para largar o Excel
Programar robôs para coletar informações online que não estão estruturadas ou disponíveis facilmente parece algo futurista, mas é uma vantagem competitiva de quem utiliza Python para analisar dados e automatizar processos. Neste artigo, apresentamos o que é a técnica web scraping, suas vantagens/desvantagens e como aplicar ela com o Python em um exemplo prático, do início ao fim.
Como usar o Python para analisar dados que não cabem no Excel?
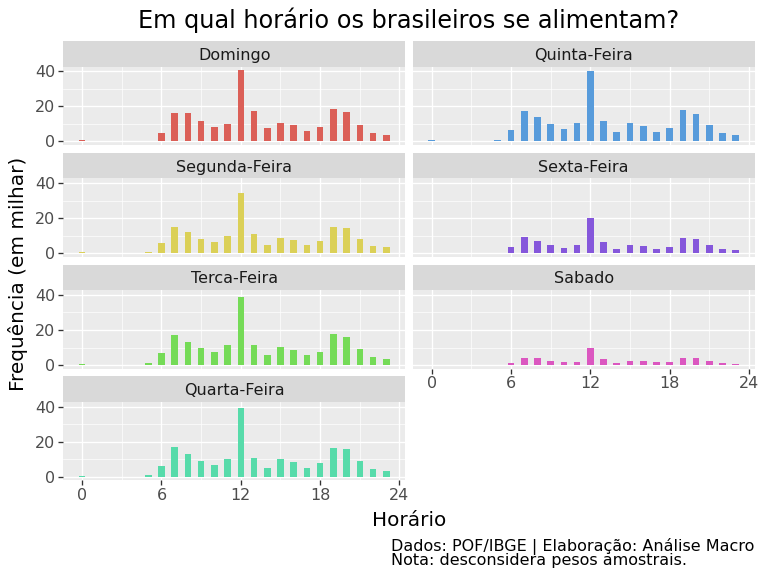
Análises de dados no Excel são limitadas a 1 milhão de linhas, o que é um grande problema numa era de Big Data. Para superar este desafio, o Python oferece diversos pacotes que lidam com grandes volumes de dados e análises complexas. Neste artigo mostramos como superar esta limitação com um exemplo prático usando dados do IBGE.

