No Hackeando o R de hoje, vamos mostrar como criar dataframes organizados quando nossos dados estão quebrados em diversas planilhas. Esse é um problema comum ao tratar de dados que são padronizados porém possuem diversas fontes, e, na falta de uma API ou outro sistema de extração automatizada, acabamos tendo que baixar uma pasta com cada um dos arquivos. Abaixo, como exemplo motivador, vamos supor que queremos extrair a localização de cada uma das estações meteorológicas automatizadas no Brasil na malha do INMET. Os dados disponíveis no site são as observações de cada ano, logo poderíamos supor que para termos todas as estações, bastaria pegar o ano mais recente de dados (2020). Apesar disso, vamos utilizar múltiplos anos, para mostrar que também podemos trabalhar com dados que pertencem a múltiplas subpastas. Em uma pasta qualquer, as planilhas têm nomes identificadores:
Além disso, como mencionado anteriormente, os dados em cada planilha são padronizados, logo as transformações aplicadas em uma também funcionarão em todas as outras. Com isso, vamos ver o formato de uma delas:
Com isso, nosso procedimento vai ser o seguinte:
- Listar todos os nomes dos arquivos relevantes e os subdiretórios onde se encontram;
- Gerar uma função que extrai os dados de um arquivo qualquer;
- Aplicar a função sobre cada um dos arquivos e agregar os resultados.
O primeiro passo é o mais simples. Primeiramente, você deve garantir que está trabalhando no diretório onde se encontram os arquivos usando setwd(), ou colocando o seu script na mesma pasta e configurando no RStudio com
Session -> Set working directory -> To source file location
Após isso, podemos listar os dados com a função list.files(). Como queremos dados que estão em subpastas, utilizamos o argumento recursive=TRUE, que indica para o R que ele deve abrir as subpastas e pegar os nomes dos arquivos que estão lá. A pasta de trabalho também possui outros arquivos, porém apenas as planilhas começam com "INMET" no nome, logo utilizamos uma expressão regular na função com o argumento pattern. A expressão abaixo começa com ^, indicando que é o começo da string, e INMET, logo são listadas as strings que começam com INMET.
arquivos = list.files(pattern = '^INMET', recursive = TRUE)
Temos então 1794 arquivos distintos. Agora, vamos utilizar o tidyverse para extraírmos os dados que queremos. Nesse exemplo, eles serão as 7 primeiras linhas de cada planilha. O código abaixo gera uma função que abre um arquivo utilizando codificação latina (para termos acentos), transforma as 2 primeiras colunas em linhas, limpa os nomes, e transforma as variáveis numéricas que eram strings devido ao uso de vírgula para decimais em números do tipo double.
library(tidyverse)
extrator = function(dados){
read_csv2(dados, locale = locale(encoding = 'latin1'), n_max=7, col_names = FALSE) %>%
pivot_wider(names_from = X1, values_from = X2) %>%
rename('Região' = `REGIÃO:`, 'UF' = `UF:`, 'Código' = `CODIGO (WMO):`,
'Latitude' = `LATITUDE:`, 'Longitude' = `LONGITUDE:`,
'Estação' = `ESTAÇÃO:`, 'Altitude' = `ALTITUDE:`) %>%
mutate(across(Latitude:Altitude, function(x) as.numeric(gsub(',', '.', x))))
}
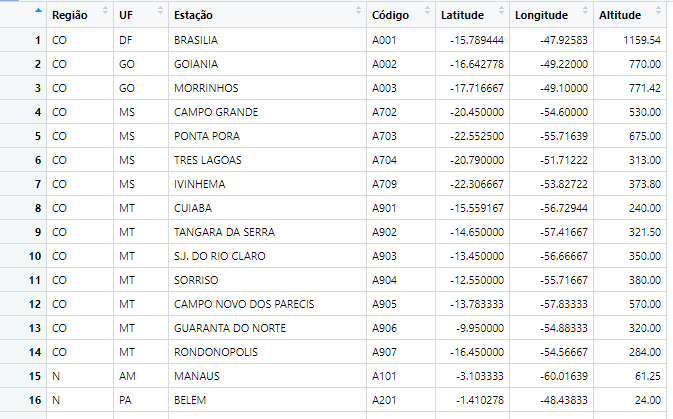
De posse da função, nos resta apenas aplicá-la sobre cada um dos arquivos. Para fazer isso, utilizaremos a função map() do pacote purrr, incluída no tidyverse. Como queremos gerar um único dataframe com cada um dos dados, e nossa função gera objetos com colunas de mesmo nome, iremos utilizar a função map_dfr, que aplica nossa função extrator() sobre cada objeto da lista arquivos, e os agrega linha por linha. É importante notar que a função será demorada, logo é bom testar com um conjunto pequeno antes de rodar sobre a lista completa. Ademais, como podemos ter repetição entre as estações, terminamos nosso código com a função unique(), que remove linhas iguais.
agregado = map_dfr(arquivos, extrator) %>% unique()