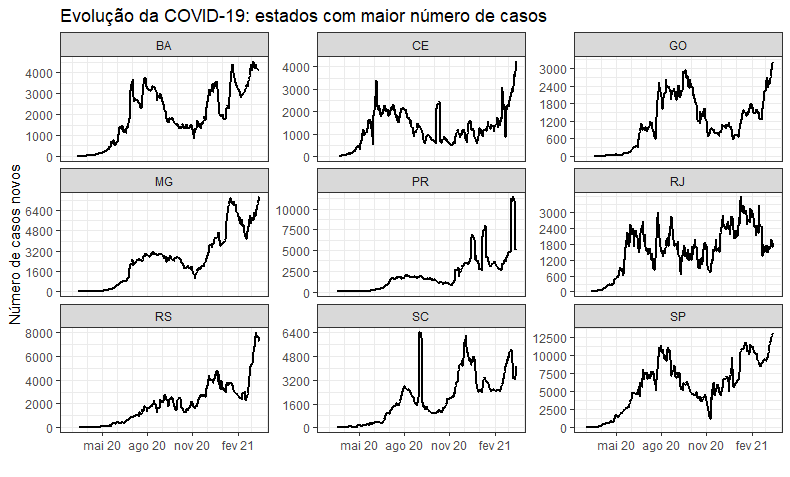
Nesse post extra, iremos mostrar outra visualização dos dados de COVID-19 apresentados no Dicas de R de quarta-feira, e também fazer um breve estudo sobre o impacto do isolamento social (lockdown) sobre os casos de gripe que apresentam SRAG, dado que essa doença possui meios de transmissão semelhantes aos do coronavírus. Inicialmente, vamos mostrar como a análise de números de casos feita no post pode ser separada em estados:
dados_covid <- vroom::vroom("https://data.brasil.io/dataset/covid19/caso_full.csv.gz")
library(tidyverse)
library(RcppRoll)
library(ggplot2)
library(ggthemes)
escalas <- function(x) {
if (max(x) > 10000) {
seq(0, 12500, 2500)
} else if (max(x) > 8000) {
seq(0, 10000, 2000)
} else if (max(x) > 6000) {
seq(0, 8000, 1600)
} else if (max(x) > 4000){
seq(0, 5000, 1000)
} else if (max(x) > 3000){
seq(0, 3000, 600)
}
}
por_estado %>%
filter(state %in% ranking[1:9]) %>%
ggplot(aes(x=date, y=value)) + geom_line(size=1.05) +
scale_y_continuous("Número de casos novos", breaks = escalas) +
scale_x_date("", breaks = "3 month", minor_breaks = "1 month", date_labels = "%b %y") +
labs(title=('Evolução da COVID-19: estados com maior número de casos')) +
theme_bw() + facet_wrap(~state, scales = "free_y")
Devemos notar que os dados desagregados possuem algumas observações que parecem ser errôneas, porém decidimos mantê-las aqui. Abaixo, um dos gráficos gerados:
Agora, vamos analisar os dados de números de casos de SRAG causados por gripe comum, conforme disponível no repositório do InfoGripe. Devido ao impacto das medidas de isolamento sobre a economia, e também sobre as fontes de renda de pessoas sob insegurança econômica, o trade-off implícito do distanciamento tem sido muito debatido, surgindo argumentos de parte da população de que o distanciamento nem mesmo seria efetivo. Como não temos dados sobre o COVID-19 sem a existência de lockdown - que seja total, parcial ou mal-aplicado -, não temos dados de controle para testar o efeito do lockdown sobre a trajetória do número de casos da doença.
Por outro lado, existem doenças que, apesar de possuírem taxas de contaminação e mortalidade menores do que o COVID-19, compartilham de seus métodos de transmissão, logo podemos inferir que, em um regime de distanciamento cujo objetivo é diminuir a transmissão do COVID-19, a redução na transmissão de outras doenças que dependem do mesmo tipo de contato evidencia a efetividade do método.
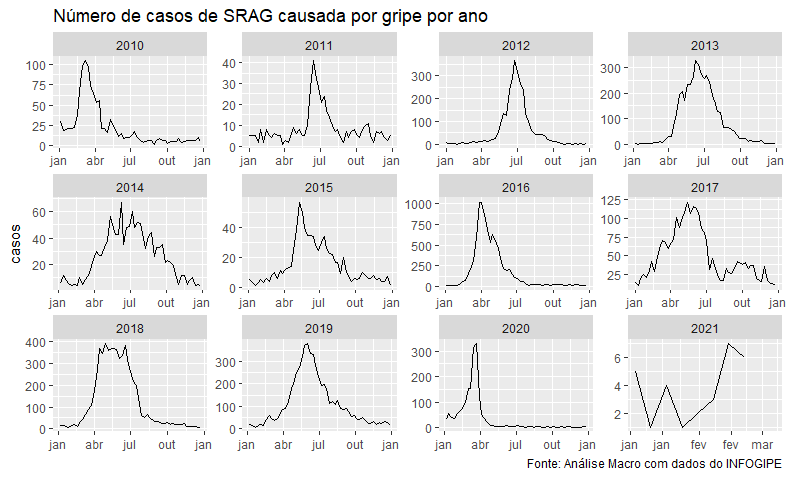
No código abaixo, vamos visualizar o número de casos ano-a-ano de gripe que apresentam SRAG:
library(ggplot2)
library(tidyverse)
infogripe <- read.csv("https://gitlab.procc.fiocruz.br/mave/repo/-/raw/master/Dados/InfoGripe/serie_temporal_com_estimativas_recentes.csv", sep = ';')
gripe_limpo <- infogripe %>% filter(Tipo == "PaÃs" & escala == "casos") %>%
select(`Ano.epidemiológico`, `Semana.epidemiológica`, `dado`,
`Casos.semanais.reportados.atÃ..a.última.atualizaÃ.Ã.o`)
srag <- gripe_limpo %>% filter(dado=="sragflu") %>%
rename(casos = Casos.semanais.reportados.atÃ..a.última.atualizaÃ.Ã.o,
ano = Ano.epidemiológico,
semana = Semana.epidemiológica) %>%
mutate(date = ano + semana/52,
date = ifelse(ano == 2014 | ano == 2020, ano+semana/53, date),
casos = as.numeric(gsub(",", ".", casos)))
srag_podado <- srag %>% filter(semana &lt;53) %>%
mutate(semana = ifelse(semana&lt;10, paste(0, semana, sep = ""), semana),
semana = gsub(" ", "", paste(semana, "01")),
date = paste(ano, semana, sep = ""),
date = as.Date(date, "%Y%W%w"))
srag_usual <- srag_podado %>% filter(ano > 2009)
ggplot(srag_usual, aes(x=date, y = casos)) + geom_line() + facet_wrap(~ano, scales = "free") +
scale_x_date(date_labels = "%b") +
labs(title = "Número de casos de SRAG causada por gripe por ano", ylab="Quantidade",
caption = "Fonte: Análise Macro com dados do INFOGIPE")+
theme(axis.title.x = element_blank())
Como podemos ver, a gripe apresenta alguma sazonalidade, e seus picos tendem a decair lentamente após o início da temporada de transmissão. É interessante notar que, no ano de 2020, há uma queda brusca no número de casos, resultado bem diferente dos anos anteriores. Além disso, o número de casos até agora em 2021 é relativamente baixo, que pode indicar uma continuação no efeito anômalo, mas também pode ser resultado de ainda não ter começado a temporada de gripe em 2021. Vamos então visualizar mais claramente os dados de 2020.
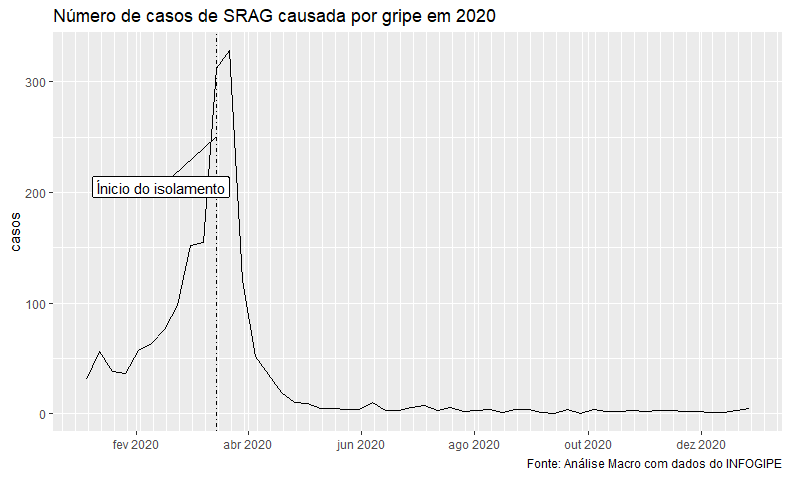
library(ggrepel)
srag_usual %>% filter(ano == 2020) %>%
ggplot(aes(x=date, y=casos))+geom_line() +
geom_vline(xintercept = as.numeric(as.Date("2020-03-15")), linetype = 4)+
scale_x_date(breaks = "2 months", minor_breaks = "1 week", date_labels = "%b %Y")+
labs(title = "Número de casos de SRAG causada por gripe em 2020", ylab="Quantidade",
caption = "Fonte: Análise Macro com dados do INFOGIPE") +
geom_label_repel(data = filter(srag_usual, date == as.Date("2020-03-15")),
label = "Ínicio do isolamento",
box.padding = 2,
aes(x=as.Date("2020-03-15"), y= 250))+
theme(axis.title.x = element_blank())
A data referenciada como início do isolamento é metade de março, época onde a maior parte dos governos estaduais decretou lockdown. Como podemos ver, o número de casos de gripe com SRAG cai bruscamente cerca de 10 dias depois, que é próximo do tempo de incubação de sintomas desse tipo para a gripe. Com isso, a análise exploratória parece indicar evidências de que utilizar o distanciamento como variável binária para controlar os casos de SRAG pode ser válido.
Faremos então a modelagem de um modelo ARDL, que tem como variáveis explicativas o lag do número de casos e a existência (ou não) de distanciamento. Os detalhes estarão disponíveis para os membros do Clube AM. A regressão final é:
| Dependent variable: | |
| casos | |
| lag(casos, 1) | 1.424*** |
| (0.039) | |
| lag(casos, 2) | -0.512*** |
| (0.046) | |
| lag(casos, 5) | 0.170*** |
| (0.046) | |
| lag(casos, 6) | -0.165*** |
| (0.039) | |
| lag(casos, 52) | 0.015 |
| (0.011) | |
| lockdown | -10.129** |
| (4.613) | |
| Constant | 5.511*** |
| (1.705) | |
| Observations | 527 |
| R2 | 0.941 |
| Adjusted R2 | 0.940 |
| Residual Std. Error | 30.076 (df = 520) |
| F Statistic | 1,370.252*** (df = 6; 520) |
| Note: | *p<0.1; **p<0.05; ***p<0.01 |
Os resultados encontrados sugerem que a existência do lockdown é estatisticamente significante, com p-valor de 0.028. O sinal do seu parâmetro é -10, logo temos evidência de que o lockdown diminui a média de casos novos semanais em mais de 10. Apesar do lag sazonal ser estatisticamente insignificante, sua exclusão aumenta o AIC e diminui a log-likelihood, logo ele é mantido aqui.
Em suma, os resultados encontrados nesse exercício sugerem que a existência do distanciamento e de medidas restritivas implica em diminuição da transmissão da gripe, que é contagiada de forma semelhante ao COVID-19. Com isso, inferimos que o lockdown é sim uma boa medida para o controle de doenças do tipo.
__________________