Para o Dicas de R dessa semana, vamos ensinar a baixar os dados sobre número de casos e óbitos de COVID de duas fontes, o repositório covid19br, e os datasets da plataforma brasil.io. Para fazer o download dos dados, basta acessar os arquivos CSV disponíveis online. Note que, no caso do brasil.io, os dados estão comprimidos, logo iremos utiilizar o pacote vroom, que baixa e extrai automaticamente as tabelas.
#não rodado
#dados_covid19br <- read.csv("https://raw.githubusercontent.com/wcota/covid19br/master/cases-brazil-states.csv")
dados_covid <- vroom::vroom("https://data.brasil.io/dataset/covid19/caso_full.csv.gz")
dados_obitos <- vroom::vroom("https://data.brasil.io/dataset/covid19/obito_cartorio.csv.gz")
Primeiramente, vamos analisar a trajetória do número de casos em território nacional. Como de costume, vamos utilizar a média móvel de 7 dias para suavizar os dados.
library(tidyverse)
library(RcppRoll)
library(ggplot2)
library(ggthemes)
dados_covid %>% filter(place_type == "state") %>%
group_by(date) %>% summarise(total=sum(new_confirmed)) %>%
mutate(casos = roll_meanr(total, n=7)) %>%
ggplot(aes(x=date, y=casos)) + geom_line(size=1.05) +
scale_x_date("", breaks = "1 month", minor_breaks = "2 weeks", date_labels = "%b %y") +
scale_y_continuous("Número de casos novos (em milhares)", breaks = seq(0, 150000, 25000), labels =
seq(0, 150, 25)) +
labs(title=('Evolução do número de casos de COVID-19 em território nacional')) +
theme_bw()
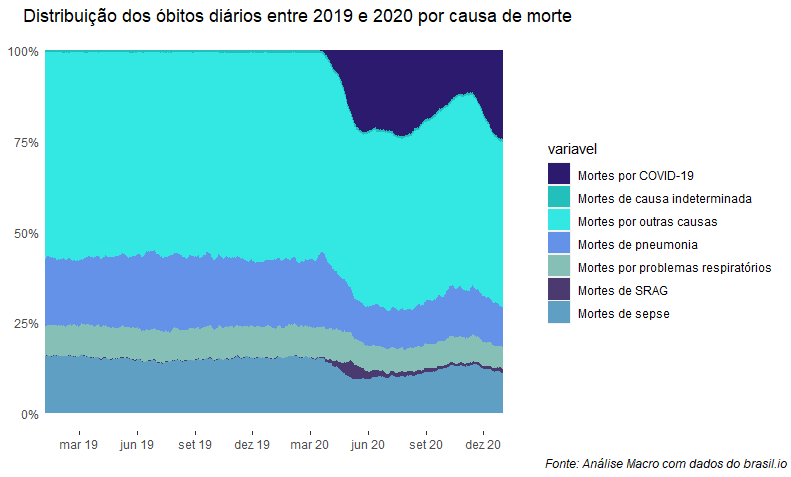
Além do número de casos, é interessante colocar em perspectiva o número de mortes causadas por COVID-19 em relação ao total de mortes do país. É claro que, além dos valores registrados, devemos ter em mente a existência de subidentificação do número de casos e óbitos, logo a proporção apresentada aqui deve ser considerada conservadora. Para fazermos a análise, vamos acessar os dados de óbitos registrados em cartórios por todo o país. Devido a limitações do dataset atualmente disponível no brasil.io, vamos restringir a visualização até 30/12/2020.
dados_2020 <- dados_obitos %>% group_by(date) %>%
summarise(deaths_covid = sum(new_deaths_covid19, na.rm = TRUE),
deaths_sars = sum(new_deaths_sars_2020, na.rm = TRUE),
deaths_others = sum(new_deaths_others_2020, na.rm = TRUE),
deaths_septicemia = sum(new_deaths_septicemia_2020, na.rm = TRUE),
deaths_pneumonia = sum(new_deaths_pneumonia_2020, na.rm = TRUE),
deaths_indeterminate = sum(new_deaths_indeterminate_2020, na.rm = TRUE),
deaths_respiratory = sum(new_deaths_respiratory_failure_2020, na.rm = TRUE),
deaths_total = sum(new_deaths_total_2020, na.rm = TRUE))
dados_2019 <- dados_obitos %>% group_by(date) %>%
summarise(deaths_covid = 0,
deaths_sars = sum(new_deaths_sars_2019, na.rm = TRUE),
deaths_others = sum(new_deaths_others_2019, na.rm = TRUE),
deaths_septicemia = sum(new_deaths_septicemia_2019, na.rm = TRUE),
deaths_pneumonia = sum(new_deaths_pneumonia_2019, na.rm = TRUE),
deaths_indeterminate = sum(new_deaths_indeterminate_2019, na.rm = TRUE),
deaths_respiratory = sum(new_deaths_respiratory_failure_2019, na.rm = TRUE),
deaths_total = sum(new_deaths_total_2019, na.rm = TRUE))
agregado <- rbind(dados_2019, dados_2020) %>%
mutate(date = seq(from = as.Date("2019-01-01"), to = as.Date("2021-01-01"), by = 'day'))
agregado %>% select(-deaths_total) %>%
mutate(across(-date, function(x) roll_meanr(x, n=7))) %>%
pivot_longer(-date, names_to = "variavel", values_to = "valor") %>%
ggplot(aes(x=date, y=valor, fill = variavel))+
geom_col(position = "fill") +
scale_y_continuous(labels = scales::percent)+
scale_fill_manual(values = c("#2b1a6e", "#22bfbb", "#33e8e2",
"#6492e8", "#86bfb6", "#4b3a70",
"#609fc4"),
labels = c("Mortes por COVID-19", "Mortes de causa indeterminada",
"Mortes por outras causas", "Mortes de pneumonia",
"Mortes por problemas respiratórios",
"Mortes de SRAG", "Mortes de sepse")) +
scale_x_date(date_breaks = "3 months", date_labels = "%b %y") +
labs(title = "Distribuição dos óbitos diários entre 2019 e 2020 por causa de morte",
caption = "Fonte: Análise Macro com dados do brasil.io") +
theme(axis.title.x = element_blank(),
axis.title.y = element_blank(),
panel.grid.major = element_blank(),
panel.grid.minor = element_blank(),
panel.background = element_blank(),
plot.caption = element_text(hjust = 2, face= "italic"),
axis.text.y = element_text(margin = margin(t = 0, r = -15, b = 0, l = 0)),
axis.ticks.y = element_blank()
)