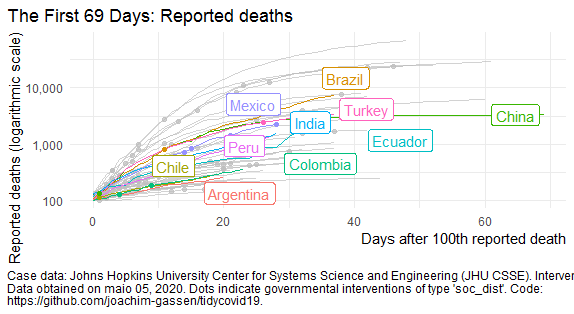
O pacote tidycovid19, disponível no github, permite customizar a visualização dos dados de Covid-19 de forma bastante simples. Veja abaixo um exemplo.
library(tidyverse)
library(tidycovid19)
covid19_dta <- download_merged_data(silent = TRUE, cached = TRUE)
plot_covid19_spread(
covid19_dta, type = "deaths", min_cases = 100, min_by_ctry_obs = 0,
edate_cutoff = 69, per_capita = FALSE, log_scale = TRUE,
cumulative = TRUE, change_ave = 7,
highlight = c('BRA', 'ARG', 'CHL', 'CHN', 'COL',
'ECU', 'IND', 'MEX', 'PER', 'PRY', 'TUR', 'URY'),
intervention = c("soc_dist")
)
(*) Aprenda R em nosso Curso de Introdução ao R para Análise de Dados.